Performing accurate species-level bacterial identification with nanopore sequencing
16S 시퀀싱은 미생물 동정에 가장 널리 사용되는 방법으로, 식품 안전, 환경 및 보존 모니터링, 병원체 탐지, 임상 미생물학 등 다양한 분야에서 활용됩니다.
16S rRNA 유전자는 약 1.5 kb 길이이며, 보존된 서열 사이에 9개의 가변 영역(V1–V9)이 존재합니다.
기존 short-read 시퀀싱 기술은 read 길이가 짧아 전체 유전자를 커버하지 못하고, 주로 일부 구간(V3–V4, V4–V5 등)만을 시퀀싱하기 때문에 종 수준 식별의 정확도가 제한적입니다.
그러나 나노포어 시퀀싱은 한 번의 read로 V1–V9 전체 영역을 포함하는 long read를 생성할 수 있어 복합균주(polymicrobial) 샘플에서도 더 높은 분류학적 해상도로 정확한 종 수준 식별이 가능합니다.

이 워크플로우는 다음과 같은 방식으로 진행됩니다:
- 16S rRNA 유전자를 전용 프라이머로 PCR 증폭한 뒤, 이를 나노포어 long read 기술로 시퀀싱하여 빠르고 비용 효율적인 species-level 식별이 가능하도록 합니다.
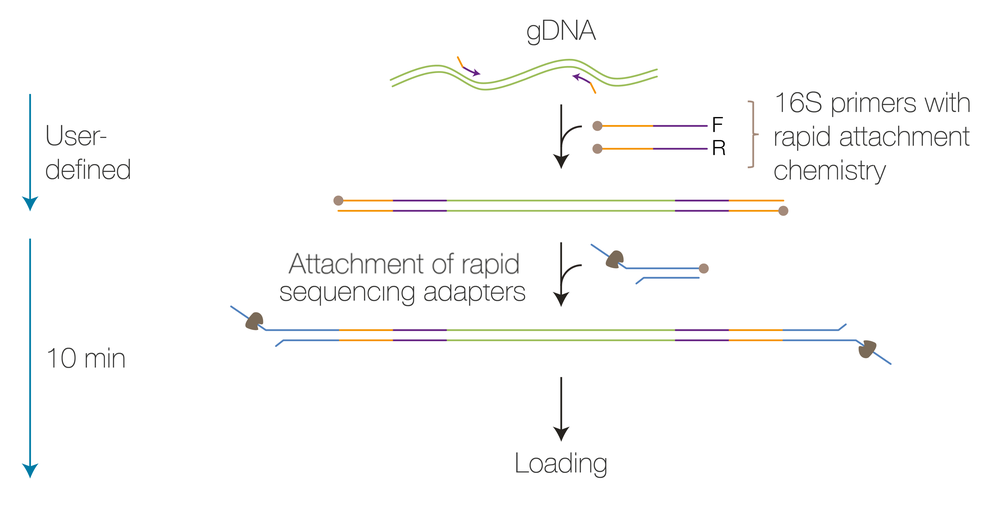
https://nanoporetech.com/document/rapid-sequencing-DNA-16s-barcoding-kit-v14-sqk-16114-24
'나노포어 어플리케이션 > 공식 워크플로우' 카테고리의 다른 글
| 메타지놈 시퀀싱을 활용한 호흡기 병원체의 빠른 식별 (0) | 2025.03.25 |
|---|---|
| AAV Genome QC with Nanopore Sequencing (0) | 2025.03.24 |
| 나노포어를 활용한 빠르고 정확한 플라스미드 전체 서열 분석 (0) | 2025.03.24 |
| Nanopore-only microbial isolate sequencing solution (NO-MISS) (0) | 2025.03.24 |
| Direct RNA Sequencing (modifications with PCR-free) (0) | 2025.03.21 |



