A systematic benchmark of Nanopore long-read RNA sequencing for transcript-level analysis in human cell lines
나노포어 롱리드 RNA 시퀀싱 기술이 사람 세포주(human cell lines) 전사체 분석에 얼마나 유용한지 체계적으로 평가했습니다.
나노포어 RNA 시퀀싱의 주요 장점
- 주요 이소폼(isoform) 식별의 정확성
짧은 리드 기반 RNA 시퀀싱에서는 주요 이소폼이 과대추정되는 경향이 있지만, 나노포어 롱리드 RNA 시퀀싱은 기술에 의존하지 않고 주요 이소폼을 안정적으로 식별할 수 있습니다. 이는 정확한 전사체 구조 분석에 큰 도움이 됩니다. - 반복서열 기반 새로운 전사체 탐지
SG-NEx 데이터 분석 결과, 나노포어 RNA 시퀀싱을 통해 기존 주석(annotation)에는 없는 반복 서열 기반의 새로운 전사체를 발견할 수 있었습니다. 특히 초기 배아 세포나 반복성이 높은 종 연구에 매우 유리한 기술임이 확인되었습니다. - RNA 변형(m6A 등) 분석 가능성
나노포어는 direct RNA 시퀀싱을 통해 RNA 분자 자체를 직접 읽을 수 있기 때문에, m6A와 같은 RNA 변형(modification)을 별도의 추가 과정 없이 직접 탐지할 수 있습니다. 이는 기존 cDNA 기반 기술과 차별화되는 중요한 강점입니다. - 오류 보정 후에도 높은 정량 신뢰성 확보
비록 나노포어 시퀀싱은 PacBio나 Illumina에 비해 상대적으로 높은 오류율을 가지지만, 오류 교정 및 스플라이스 정렬 보정 기법(Bambu, Flair, NanoSplicer 등)을 적용하면, 전사체 발현 정량 결과가 매우 일관성 있게 유지될 수 있습니다. - 다양한 프로토콜과 세포주에 대한 높은 적용성
이번 연구에서는 다섯 가지 RNA-seq 프로토콜과 일곱 개의 인간 세포주를 대상으로 실험했으며, 모든 조건에서 나노포어 데이터는 전사체 정량화, 새로운 전사체 발견, 융합 유전자 탐지 등에 안정적으로 활용될 수 있음을 보여주었습니다.
연구 목적
유전자 하나는 다양한 형태의 RNA(아이소폼, isoform)를 만들어낼 수 있습니다. 하지만 기존의 짧은 읽기(short-read) RNA 시퀀싱은
- 아이소폼을 완전히 구별하기 어렵고, 복잡한 전사 변화를 포착하는 데 한계가 있었습니다.
이번 연구는 다양한 RNA 시퀀싱 방법을 비교하여, 롱리드 시퀀싱이 아이소폼 분석에 얼마나 뛰어난지를 평가했습니다.
실험 방법
- 7개 인간 세포주를 대상으로 분석
- 비교한 RNA 시퀀싱 방법:
- 짧은 읽기 기반 cDNA 시퀀싱
- 나노포어 direct RNA 시퀀싱
- 나노포어 direct cDNA 시퀀싱 (PCR 없이)
- PCR 증폭된 cDNA 시퀀싱
- PacBio Iso-Seq 롱리드 시퀀싱
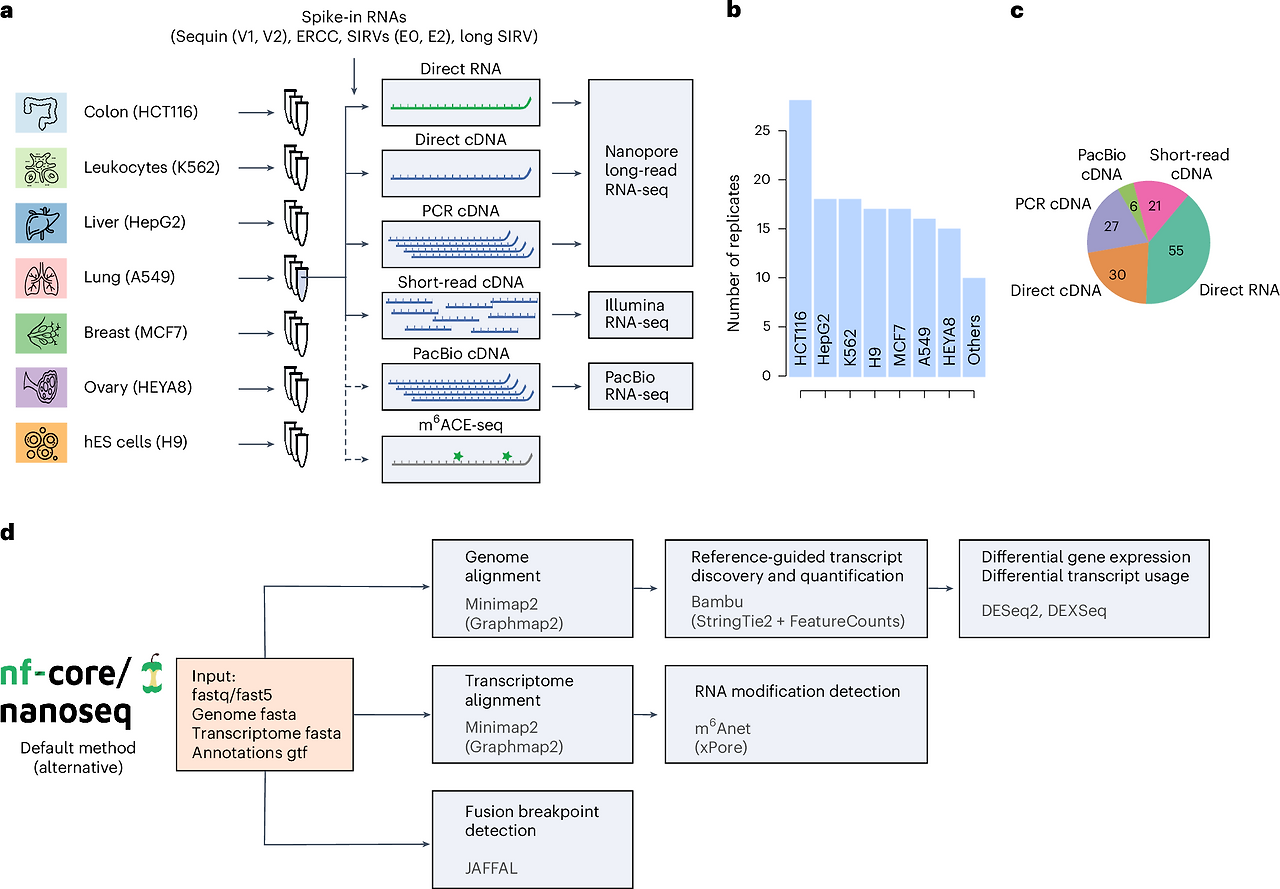
- 추가로:
- 스파이크인 외부 기준 물질 사용
- RNA 메틸화(m6A) 데이터 분석
주요 결과
- 롱리드 시퀀싱은 전사체의 전체 구조를 포착할 수 있어, 짧은 읽기보다 아이소폼(대체 전사체) 분석 정확도가 훨씬 높았습니다.
- 나노포어 direct RNA 시퀀싱은
- 메틸화(m6A) 등 RNA 수정까지 탐지할 수 있어
- 전사체의 구조 + 수정 정보까지 한 번에 분석할 수 있었습니다.
- 새로운 전사체(novel transcripts)나 융합 전사체(fusion transcripts) 탐지에서도 롱리드 기술이 뛰어난 성능을 보였습니다.
- 이번 연구를 통해 생성된 데이터 세트는 앞으로 새로운 RNA 분석 소프트웨어 개발이나 복잡한 전사체 연구에 매우 유용하게 활용될 수 있습니다.
결론
나노포어 롱리드 RNA 시퀀싱은 짧은 읽기 기술로는 확인하기 어려운
- 대체 스플라이싱(Alternative Splicing)
- 새로운 전사체(New Transcripts)
- RNA 수정(m6A methylation) 을 포괄적으로 탐지할 수 있다는 점에서 큰 가능성을 보여주었습니다.
앞으로 전사체 수준의 정밀 분석이 필요한 연구나, 신약 개발, 희귀 질환 연구에서도 롱리드 RNA 시퀀싱이 핵심 기술이 될 것으로 기대됩니다.